| UHH > Fachbereich Biologie > Lehrmaterial > Biochemie zum Ansehen > Virologie | |
Kleine Organismen müssen mit ihren Kräften und Vorräten haushalten. Das gilt besonders für Viren - wenn man Viren als Organismen ansieht. Dazu gibt es eine Reihe von minimalistichen Ansätzen, von nackten Genomen in Viroiden bis hin zu mehrschichtigen Proteinhüllen, die den genetischen Bauplan zusammen mit einigen Enzymen einschließen. Wenn das Genom mit möglichst wenig Protein geschützt werden soll (wenig im Sinne von Codierungsaufwand), bietet sich eine Lösung an, bei der nur ein Protein als sich wiederholender Baustein für eine Kapsel benutzt wird. Diese Methode wird beispielsweise von den ikosaedrischen Viren angewendet: ihr Capsid besteht aus 60 gleichen Einheiten, deren Symmetrieeigenschaften die Anordnung zum Ikosaeder erlaubt. Die zugrundeliegenden Symmetrieanforderungen werden hier in dem Script "Symmetriebeziehungen in ikosaedrischen Capsiden" erläutert.
Viren verschiedener taxonomischer Gruppen sind nach diesem Prinzip gebaut. Das Genom kann von einsträngiger oder doppelsträngiger Nucleinsäure codiert sein, entweder RNA oder DNA. Die Proteinuntereinheiten können aus einer, zwei, drei oder vier Ketten bestehen. Mehrere Ketten können nach der Synthese durch Spaltung aus einem Protein entstehen, und Proteinketten können verschieden gefaltet werden, wie es Symmetrieanforderungen bezüglich ihrer Lage zu Symmetrieachsen erfordern. Ähnlichkeiten im Faltungsmuster verschiedener Einzelproteine lassen auf einen gemeinsamen Vorfahren schließen.
Das Ikosaedergerüst kann mit mehr als 60 Untereinheiten aus Protein bestückt sein, sodaß komplexere Capside entstehen. Eine Übersicht ikosaedrischer Viren aller Symmetrieklassen (soweit ihre Struktur bei etwa atomarar Auflösung bekannt ist) findet sich in der Internet-Datenbank VIPER (Virus Particle Explorer).
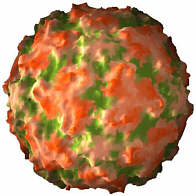
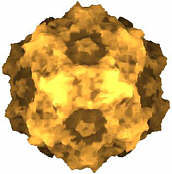
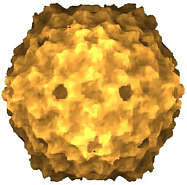
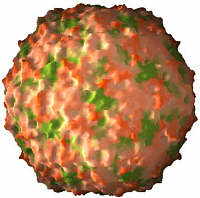
Unter den 'kleinen' ikosaedrischen Viren gibt es wohlbekannte Krankheitserreger von Menschen und Tieren, die z.B. Poliomyelitis, Schnupfen, Leberentzündung, Maul- und Klauenseuche oder eine Reihe von Darmerkrankungen hervorrufen. Pflanzenpathogene wie das Gelbfleckenvirus von Reis können Landstrichweise eine Jahresernte vernichten. Bei Insektenviren und Bakteriophagen gibt es das gleiche Bauprinzip. In der folgenden Bildleiste sind einige dieser Viren abgebildet, die Einfärbung der Bilder soll lediglich die Oberflächenbeschaffenheit modellieren. Die Orientierng (und relative Größe) sind jeweils gleich, sodaß die Symmetrieachsen leicht zu finden sind.
 |
 |
 |
 |
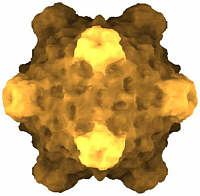 |
| human rhinovirus | porcine parvovirus | rice yellow mottle virus | cricket paralysis virus | bacteriophage phiX174 |
| Bilder © VIPER (http://mmtsb.scripps.edu/viper/) | ||||
Picornaviren haben große Bedeutung in Medizin, Gesundheitsvorsorge und Ökonomie, daher wurden sie in den letzten Jahrzehnten intensiv erforscht. Daraus resultierte z.B. das globale Polio-Eliminierungsprogramm der Weltgesundheitsorganisation, aber auch ein großer Teil von Strukturinformation seit den 1980er Jahren. Man kennt daher heute auf molekularer Ebene die Wechselwirkung von Viren mit zellulären Rezeptoren, neutralisierenden Antikörpern und Antibiotica. Die Strukturen von viruscodierten Enzymen sind auch publiziert.
In den hier folgenden Jsmol-Scripten (basierend auf früheren Chime-Scripten) sind keine Modelle von ganzen Viren zu sehen. Wenn die Viren auch 'klein' genannt werden, die Strukturdatenfiles für ganze Capside sind es nicht. Weder Chime noch Jsmol wurden mit dem Ziel programmiert, mit Files von >30MB umzugehen: ein Bildaufbau in raumfüllender Darstellung bräuchte mehrere Minuten. Auch die Darstellung 'nur' aller Liganden um eine Symmetrieachse läßt die Bilder rucken.
 |
Scripte über Poliovirus (wenn kein Link, ist es in Vorbereitung ;-)):
|
DL Caspar & A Klug, Physical principles in the construction of regular viruses, Cold Spring Harbor Symp. Quant. Biol. 27 (1962) 1-24
MG Rossmann & JE Johnson, Icosahedral RNA virus structure, Annu. Rev. Biochem. 58 (1989) 533-573
JE Johnson & JA Speir, Quasi-equivalent viruses: a paradigm for protein assemblies, J. Mol. Biol. 269 (1997) 665-675
TS Baker et al, Adding the third dimension to virus life cycles: three-dimensional reconstruction of icosahedral viruses from cryo-electron microscopy, Microbiol. Mol. Biol. Rev. 63 (1999) 862-922
SC Harrison, The familiar and the unexpected in structures of icosahedral viruses, Curr. Opin. Struct. Biol. 11 (2001) 195-199
VS Reddy et al, Virus Particle Explorer (VIPER), a Website for virus capsid structures and their computational analysis, J. Virol. 75 (2001) 11943-11947
N Verdaguer et al, Structure of human rhinovirus serotype 2 (HRV2), J. Mol. Biol. 300 (2000) 1179-1194)
AA Simpson et al, The structure of porcine parvovirus: comparison with related viruses, J. Mol. Biol. 315 (2002) 1189-1198
C Qu et al, 3D domain swapping modulates the stability of members of an icosahedral virus group, Structure 8 (2000) 1095-1103
J Tate et al, The crystal structure of cricket paralysis virus: the first view of a new virus family, Nature Struct. Biol. 6 (1999) 765-774
R McKenna et al, Analysis of the single-stranded DNA bacteriophage phiX174, refined at a resolution of 3.0 Å, J. Mol. Biol. 237 (1994) 517-543