Poliovirus RNA-Polymerase 3D
Poliovirus codiert seine eigene RNA-Polymerase (RdRP, RNA-dependent RNA Polymerase). Die Struktur dieses Enzyms gleicht der der meisten anderen Nucleinsäure-Polymerasen und erinnert an eine halboffene Hand. Dementsprechend werden die Domänen der Enyme benannt: es gibt einen Handteller
mit einem kräftigen Daumen
und eine Finger-Domäne
. Eine Eigenart der Poliovirus Polymerase sind aminoterminale Strukturen, die es in anderen Polymerasen nicht gibt
. Strukturverwandte Domänen haben auch ähnliche Funktionen, Finger und Daumen binden das Subsrtrat (Primer-RNA und Matritzenstrang) und die Handfläche besorgt die Phosphoryltransfer-Reaktion.
In diesem Strukturmodell gibt es mehrere lose Enden
: In den Bildern der Röntgenstrukturanalyse gibt es Bereiche mit unzulänglicher Elektronendichte, sodaß die entsprechenden Aminosäuren nicht exakt lokalisiert werden konnten (das Molekül kann immer mit der Maus gedreht werden, um es von allen Seiten zu betrachten!)
Die Handtellerdomäne besteht aus Motiven, die auch in anderen Polymerasen gefunden werden.
Met225 - Leu241Am Ende der kurzen Helix in Motiv A gibt es das in den Polymerasestrukturen konservierte Ser240 . Es bildet Wasserstoffbrücken zu den carbonyl-Sauerstoffatomen von Gly236 und Tyr237 und stabilisiert damit diese Schleife.
Cys290 - Thr312
Leu318 - His336
Asp339 - Met354
Glu369 - Ala380
In Motiv A gibt es auch ein komplett konserviertes Aspartat - in allen Polymerasen koordiniert diese Aminosäure ein katalytisch notwendiges Metallatom .
Eine weitere Gruppe von Aminosäuren ist in RNA-abhängigen RNA-Polymerasen konserviert: Asp238 in Motiv A und Asn297 in Motiv B . An genau diesen Positionen befinden sich in RNA-abhängigen DNA-Polymerasen aromatische Aminosäuren (Tyr und Phe in HIV Reverser Transcriptase). Diese Aminosäuren dienen daher wohl der Unterscheidung zwichen Ribo- und Desoxyribonucleotiden bei der Polymerisation der Nucleinsäure.
In der beta-Schleife in Motiv C gibt es weitere katalytisch wirksame Aspartate . Mutation von Asp233 oder Asp328 inaktiviert die Polymerase, ein Wechsel von Asp329 zu Asn ändert die Metallbindungsspezifität .
Motiv E gibt es nur in RNA-abhängigen Polymerasen. Aminosäuren aus diesem Motiv wechselwirken mit dem wachsenden Initialisierungssstrang. Das Motiv stellt außerdem die Verbindung zur Daumen-Domäne durch die Bildung eines Falrblattes her .
In diesem Modell der Polio RdRP ist ein großer Teil der Finger-Domäne nicht sichtbar . Einen Eindruck von der Form des gesamten Enzyms gibt jedoch ein Vergleich mit einer strukturell eng verwandten RdRP (aus rabbit hemorrhagic disease virus), die allerdings 40 Aminosäuren größer als das Polio-Enzym ist . In der raumfüllenden Ansicht ist die 'Hand' geschlossen, nur ein Loch läßt die Substrat-RNA passieren. Der bekannte Teil der Polio-RdRP nimmt das gleiche Volumen ein, mit Ausnahme der aminoterminalen Sequenz (12 - 24) , die den Kanal im Enzym blockiert. Das mag ein Artefakt sein, da im Kristallisationsexperiment keine RNA enthalten war.
Hochgereinigte Polio-RdRP kann in vitro initialisierte RNA-Synthese alleine ausführen. Innerhalb von Zellen oligomerisiert das Enzym jedoch zu komplexen Netzwerken, die mit Membranen verbunden sind. In der Polymerase wurden Kontaktbereiche an der Oberfläche gefunden, die überkreuz angeordnet sind. Kontaktbereich I wird durch 23 Aminosäuren an gegenüberliegenden Seiten des Mooleküls gebildet, die eine Aneinanderlagerung zu (theoretisch unbegrenzt langen) Ketten ermöglicht. Der andere Kontaktbereich (II) erlaubt in der anderen Richtung eine Kettenbildung, sodaß ein zweidimensionales Netz entstehen kann. In dieser Richtung ist ein Metallion beteiligt. Die physische Nähe der Polymerasemoleküle zueinender erlaubt eine effektiv gekoppelte Synthese von plus- und minus-Strängen der viralen RNA sowie Rekombinationsereignisse.
Die 3D-Polymerase von Polio ist selbst kein membrangebundenes Enzym. Die Anheftung an Membranen erfolgt mithilfe eines anderen Virenproteins, 3AB. 3AB wird durch Aminosäuren der Polio-Polymerase gebunden , die senkrecht zu der Ebene der das Netzwerk formenden Kontaktflächen angeordnet sind.
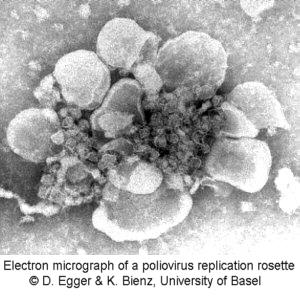
Das elektronenmikroskopische Bild eines isolierten Replikationskomplexes zeigt den intrazellulären Arbeitsplatz der Polymerase. Diese charakteristischen Strukturen werden Rosette genannt.
Zurück zum .
Literatur:
JL Hansen et al, Structure of the RNA-dependent RNA polymerase of poliovirus, Structure 5 (1997) 1109-1122
DW Gohara et al, Poliovirus RNA-dependent RNA polymerase, J. Biol. Chem. 275 (2000) 25523-25532
HB Pathak et al, Structure-function relationships of the RNA-dependent RNA polymerase from poliovirus (3Dpol), J. Biol. Chem. 277 (2002) 31551-31562
JM Lyle et al, Visualization and functional analysis of RNA-dependent RNA polymerase lattices, Science 296 (2002) 2218-2222
D Egger & K Bienz, Recombination of poliovirus RNA proceeds in mixed replication complexes originating from distinct replication start sites, J. Virol. 76 (2002) 10960-10971
KKS Ng et al, Crystal structures of active and inactive conformations of a calciviral RNA-dependent RNA polymerase, J. Biol. Chem. 277 (2002) 1381-1387
 Biochemie zum Ansehen
Biochemie zum Ansehen 