Protomer des Poliovirus-Capsids
Die PDB-Strukturdaten stammen aus 1hxs.unique; sie mußten angepaßt werden, damit Chime/Jsmol damit umgehen können. Auch in komprimierter Form ist der File 1MB groß, deshalb bitte Geduld beim Laden.
Das Capsid von Poliovirus besteht aus 60 Untereinheiten zu je vier Proteinketten. Hier ist eine Untereinheit (oder Protomer) mit seinen Proteinen gezeigt:
-
VP1
(302 Aminosäuren)
-
VP2
(272 Aminosäuren)
-
VP3
(238 Aminosäuren)
-
VP4
(68 Aminosäuren)
VP4 wird zusammen mit VP2 synthetisiert und erst abgespalten, wenn der Komplex (VP0) in das Capsid eingebaut wird. In dem Datenbankeintrag in der PDB wird der Myristylrest am Aminoende von VP4 als Nummer 1 gezählt .
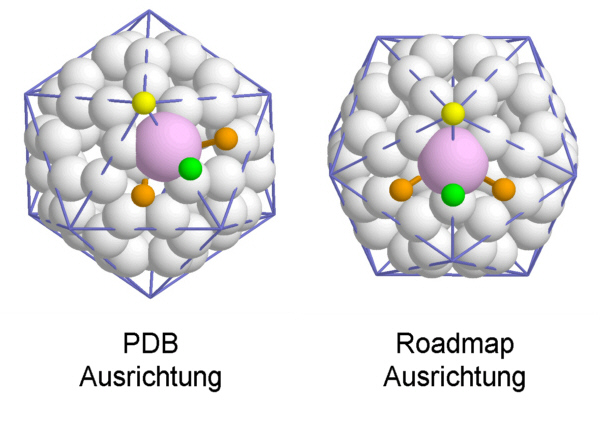
Welche der viralen Proteine ein Protomer bilden, ist eine willkürliche Auswahl. Hier wird die Wahl in Analogie zu Pflanzenviren mit ähnlicher Struktur getroffen. In der PDB sind die Daten für ein Protomer in der links gezeigten Konfiguration abgelegt : Eine fünfzählige Achse bildet den Nordpol und eine dreizählige Achse zeigt nach Süden. Man kann jedoch genausogut die 'Straßenkarten' (Roadmap) - Ansicht wählen, wenn man das rechts liegende VP3 einbezieht:
Die Faltungsschemata der drei großen Proteine sind in etwa gleich: Ein großes beta-Faltblatt aus acht antiparallelen Strängen ist zu einem verzerrten Faß gewunden. In zweidimensionaler Darstellung ist deutlich ein Mäander-Motiv zu erkennen.
VP1 beta-Faltblatt
VP1 Helices
VP2
Proteinkette
VP2
beta-Faltblatt
VP2
Helices
VP3 beta-Faltblatt
VP3 Helices
Bei genauerem Hinsehen stellt man fest, daß die beta-Fässer nicht ganz geschlossen sind. Sie sollten besser als beta-Sandwich bezeichnet werden.
VP4
Proteinkette
VP4
beta-Faltblatt (mit VP1)
VP4
Helices
Die Strukturen von VP1 bis VP3 können zu einem gemeinsamen Muster abstrahiert werden. In der Literatur findet man folgende Bezeichnungen für Stränge und Schlaufen:
Das Gewirr der Verbindungslinien der Calpha-Atome deutet schon auf enge Kontakte der Proteine innerhalb eines Protomers hin. Deutlich wird es bei Betrachtung im raumfüllenden Modus . Die Lücken an den Rändern des Protomers werden beim Zusammensetzen des Capsides von benachbarten Protomeren gefüllt.
Wechselwirkung der Protomere
Die dem hier gezeigten Proteinmodell zugrundeliegenden Daten stammen aus ganzen Viren, das Modell repräsentiert also nicht ein isoliertes Protomer in Lösung. Die Struktur muß daher im Zusammenhang mit anderen Protomeren betrachtet werden. Wechselwirkungen sind besonders im Bereich der Symmetrieachsen des Virus interessant.
Eine fünfzählige Achse
wird an der Oberfläche des Partikels von VP1 umgeben. VP4 wird vollständig von anderen Proteinen überdeckt, es ist nur von innen zugänglich
. Der aminoterminale Teil von VP4 berührt mehrere andere Protomere an der Achse
. In dem Pentamer gibt es weitere Strukturelemente, die zur Stabilisierung beitragen
. Die Aminoenden von VP3 bilden zusammen ein beta-Fass
. Zusätzliche kurze beta-Faltblätter werden auf der inneren Oberfläche durch Stränge von VP1 und zwei VP4 (von demselben und einem benachbarten Protomer) gebildet
. Die Myristoylreste von VP4 formen eine Art hydrophoben Stopfens innen vor dem beta-Fass
.
Die Schlaufen von VP1 um die fünfzählige Achse erheben sich zu einem Turm über die Oberfläche des Virus
. Die geladenen Aminosäuren dieser Schlaufen generieren elektrostatische Kräfte
, während hydrophobe Aminosäuren
die Protomere durch van der Waals-Kräfte binden. Im Schnittbild des Pentamers sind um den VP1-Turm tiefe Furchen zu sehen
, die in der Literatur Canyon genannt werden.
Ein Blick auf eine dreizählige Achse zeigt auf der äußeren Oberfläche alternierend VP2 und VP3. Innen sind alle VPs zu sehen, wobei sie sich gegenseitig durchdringen. Eine Besonderheit der Struktur gibt Zusammenhalt um diese Achse. Eine Seite des beta-Sandwich in VP3 wird durch das 'Extra'-Faltblatt von VP2 (Aminosäuren 14 - 25) des Nachbarprotomers fortgesetzt . Um einen weiteren Strang wird das Faltblatt durch Aminosäuren von VP1 des ersten Protomers erweitert , sodaß die Protomere ineinandergesteckt sind (ein guter Grund, das beta-Faß von VP3 zu einem Sandwich zu öffnen). Dieses Strukturmerkmal findet sich natürlich in allen drei Grenzflächen um die Achse .
Protomer-Wechselwirkungen um die zweizähligen Achsen ergeben sich zwangsläufig aus denen um die dreizähligen Achsen . Zusätzlich liegen sich Helices von VP2 genau an der Achsenposition gegenüber. Ihre Aminosäuren haben engen Kontakt .
Zurück zum .
Literatur:
JM Hogle et al, Three-dimensional structure of poliovirus at 2.9 Å resolution, Science 229 (1985) 1358-1365
ST Miller et al, Ab initio phasing of high-symmetry macromolecular complexes: successful phasing of authentic poliovirus data to 3.0 Å resolution, J. Mol. Biol. 307 (2001) 499-512
C Zhang & SH Kim, A comprehensive analysis of the Greek key motifs in protein beta-barrels and beta-sandwiches, Proteins Struct. Funct. Genet. 40 (2000) 409-419
 Biochemie zum Ansehen
Biochemie zum Ansehen 