RecBCD: Reparatur von DNA-Strangbrüchen
Ein Schaden der DNA ist für jede lebende Zelle fatal, wenn er nicht ohne Informationsverlust repariert wird. In Eubakterien werden Doppelstrangbrüche über den homologen Reparationsweg repariert. Dabei wird die gebrochene DNA durch teilweises Verdauen so vorbereitet, daß der Punkt für eine Religation erreicht wird. Die Anfangsschritte werden durch RecBCD ausgeführt, ein Multienzymkomplex mit der Funktion von Nucleasen und Helicasen. Das Enzym verdaut beide Stränge von der Bruchstelle aus, bis es auf eine Chi-Sequenz (Crossover hotspot instigator) stößt. Von hier aus wird nur noch der 5' -> 3'-Strang der DNA weiter verdaut, der andere Strang bleibt intakt und bindet RecA-Protein. Das RecA-Filament initiiert eine Rekombination mit einem anderen auf diese Weise vorbereiteten DNA-Ende. Die für die Vorbereitungsreaktion nötige Energie stammt aus der Hydrolyse von ATP.
RecBCD ist ein Heterotrimer von 330 kDa. RecB funktioniert sowohl als 3'-5' Helicase als auch als multifunktionelle Nuclease. RecC erkennt die Chi-Sequenz 5'-GCTGGTGG-3' und limitiert daraufhin die RecB-Nucleaseaktivität auf den 5'-3'-Strang. RecD arbeitet als 5'-3'-Helicase.
Die in diesem Experiment benutzte Nucleinsäure wurde als selbskomplementäres 43mer hergestellt . In der Röntgenkristallstruktur sind die zentralen Basen T20-T-C-C-C24 nicht sichtbar. Die Basen 5-19 paaren zu 25-39 und bilden eine reguläre B-DNA, die übrigen Basenpaare sind schon getrennt und in dem Proteinkomplex verborgen . Der Doppelstrangteil der DNA wird von einem Arm des RecB-Proteins gehalten (mit der Maus kann man den Komplex drehen und dann auch die Kanäle mit der Einzelstrang-DNA finden!)
Der Helicase-Teil von RecB
ist strukturell ähnlich zu der Helicase-Überfamilie 1.
Domäne 1A
enthält ein zentrales paralleles beta-Faltblatt mit einer zu RecA ähnlichen Struktur. Sowohl ein
als auch ein
Motiv sind vorhanden. Die Walker A-Struktur Ala-x-x-x-x-Gly-Lys-Thr
umschließt die Phosphatgruppe eines Nucleotides und hält es dadurch fest. Neben Walker B (hier die Sequenz Val-Ala-Met-Ile-Asp)
ist Glu385 die katalytische Aminosäure der ATPase. Dieser Teil von RecB
stellt also den Energielieferanten der Helicaseaktivität dar. Domäne 1B verbindet die Helicase
(hellrün) mit dem Arm, der die Doppelstrang-DNA hält
. Ein Strukturelement wie Domäne 1B ist auch in anderen Helicasen zu finden, der Arm findet sich jedoch nur in RecB. In diesem Arm sind die Aminosäuren 289 bis 304
in der Röntgenstruktur nicht sichtbar.
Die Struktur von Domäne 2A
entspricht wieder dem Muster der Helicase-Überfamilie. Sie bindet das 3'-Ende der Einzelstrang-DNA
; zusammen mit Domäne 1A
bildet sie den Helicase-Motor. Domäne 2B hat eine Sekundärstruktur aus Helices. Sie zeigt von dem Helicase-Motor weg und stellt die Verbindung zu RecC her
.
Von den Domänen 1A - 1B - 2A - 2B führt ein Aminosäurefaden
zu Domäne 3, der Nuclease
. Das Strukturmuster dieser Nuclease
findet sich auch in der Exonuclease des Phagen Lambda, wobei die Aminosäuren des katalytischen Zentrums gleich sind
. Ein Magnesium-Ion
ist Teil des aktiven Zentrums
.
Die Abbildung unten zeigt das Faltungsschema von RecB
ohne die helicalen Bereiche:
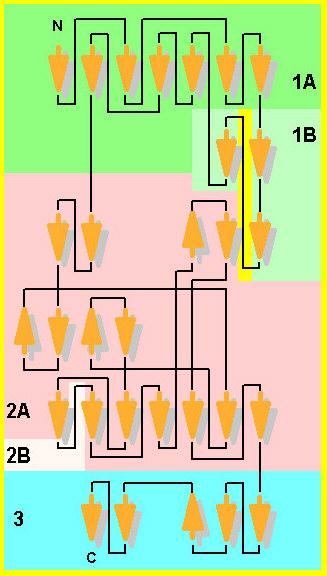
Das Faltungsschema von RecC mit seinen großen parallelsträngigen beta-Faltblättern deutet auf seine Wurzeln in der Helicase-Überfamilie ähnlich wie RecB.
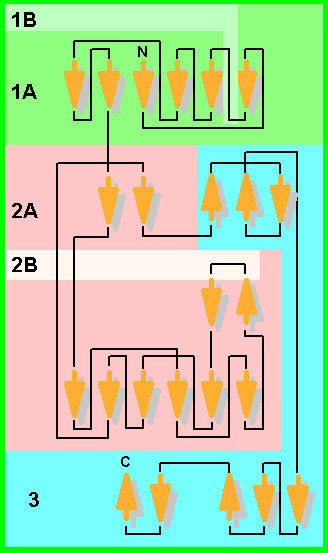
Die räumliche Anordnung der Domänen ist jedoch spezifisch für RecC. Es gibt drei große Öffnungen in der Struktur. Die zentrale Höhle wird begrenzt von den Domänen 2A und 2B . In diese Lücke stößt Domäne 2B von RecB . Die von Domäne 3 umgebene Öffnung umschließt das 5'-Ende der abgedauten DNA . Aus Domäne 3 stößt eine Nase hervor , an der die DNA aufgespalten wird . Bei einer "echten" DNA in vivo würde das 5'-Ende der Einzelstrang-DNA durch das Loch weiterführen bis zur zweiten Helicase, RecD. Das andere freie DNA-Ende (3') wäre ebenfalls länger; es würde durch das dritte Loch vorbei an dem RecB-Helicasemotor zu dem Nuclease-Teil von RecB führen (wenn Sie das Schnittbild links mit der Maus bewegen, können Sie die Kanäle im RecBCD Komplex sehen!).
Das Faltungsschema der anderen Helicase-Untereinheit, RecD , entspricht nur teilweise dem Helicase-Überfamilienschema; insbesondere fehlt Domäne 1 das große parallelsträngige Faltblatt . Die Arbeitsrichtung von RecD ist in Bezug zur Strängigkeit der DNA entgegengesetzt zu der anderen Helicase (muß sein, weil sie sonst herunterfällt); das Fehlen einiger Strukturmotive in Domäne 1 mag damit zusammenhängen.
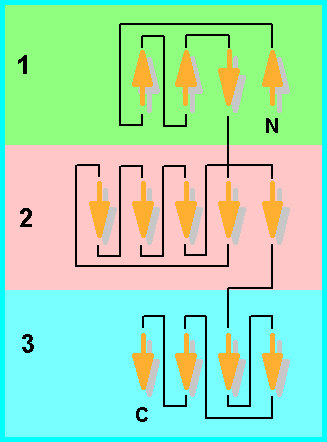
Die Domänen 2 und 3 entsprechen den Motordomänen anderer Helicasen, die ATPase-Motive Walker A und Walker B liegen in Domäne 2 .
Bei der Arbeit des RecBCD-Komplexes werden beide Einzelstrang-Enden der DNA von eigenen Helicase-Motoren angetrieben. Für die Verdauung der überschüssigen Einzelstrang-DNA gibt es aber nur eine Nuclease, Domäne 3 von RecB . Die Lage in der Nähe der Austrittpunkte der Einzelstrang-DNA ermöglicht es der Nucleasen-Domäne, sich von RecC und dem 3'-Ende der DNA abzuwenden. Sie kann dann das 5'-Ende erfassen und abbeißen.
RecC enthält die Chi erkennende Stelle, die durch Mutationsexperimente identifiziert werden konnte . Wenn diese Aminosäuren an die Erkennungssequenz 5'-GCTGGTGG-3' binden, wird die Bewegung der DNA in Richtung Nuclease angehalten. Die Helicase RecB arbeitet jedoch weiter und schiebt den Einzelstrang in einem Bogen aus dem Komplex heraus. Die Nuclease kann also nur noch den anderen Strang abdauen, sodaß schließlich die für die Religation nötigen überhängenden Enden der DNA entstehen.
Zurück zum .
Literatur:
MR Singleton et al, Crystal structure of RecBCD enzyme reveals a machine for processing DNA breaks, Nature 432 (2004) 187-193
MR Singleton & DB Wigley, Modularity and specialization in superfamily 1 and 2 helicases, J. Bacteriol. 184 (2002) 1819-1826
 Biochemie zum Ansehen
Biochemie zum Ansehen 